现况
现有的滤波器剪枝的标准是,“smaller-norm-less-important”,即认为滤波器的范数(p-norm)越小,相对应的特征图越接近于 0,于是对网络对贡献越小,那么这些滤波器可以去掉而不会严重影响网络的性能。 但作者认为这个标准要达到好的效果需要满足两个前提:
- 不同卷积核之间的范数差距要够大
- 最不重要的(范数最小的)卷积核的范数要够小
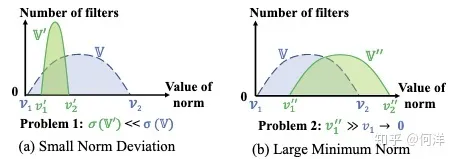 即如图 (a) 可能会导致卷积核之间的重要性难以区分,间隔区间太小,而图 (b) 剪去的卷积核的范数还是很大,即对结果的影响还是很大,删去后影响较大。
即如图 (a) 可能会导致卷积核之间的重要性难以区分,间隔区间太小,而图 (b) 剪去的卷积核的范数还是很大,即对结果的影响还是很大,删去后影响较大。
介绍
因此,作者提出了一种用几何中位数来描述卷积核的可替代性,从而进行剪枝的方案
几何中位数是对于欧几里得空间的点的中心的一个估计。我们认为滤波器也是欧氏空间中的点,于是我们可以根据计算 GM 来得到这些滤波器的“中心”,也就是他们的共同性质。如果某个滤波器接近于这个 GM,可以认为这个滤波器的信息跟其他滤波器重合,甚至是冗余的,于是我们可以去掉这个滤波器而不对网络产生大的影响。去掉它后,它的功能可以被其他滤波器代替。
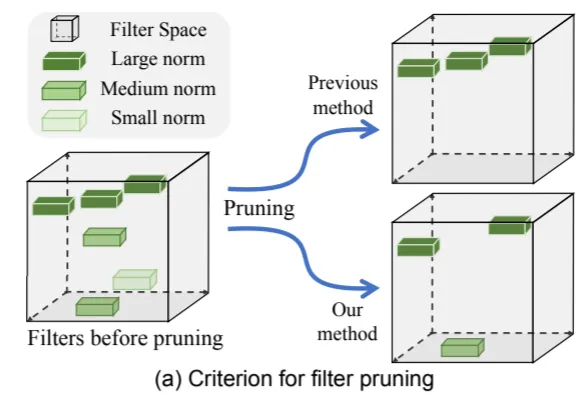 右上角即为传统方法进行剪枝,右下角为作者提出的方法(FPGM)进行剪枝
和之前看的 [[Network Compression Based on Filter Similarity Pruning for Fast Inference on Edge Devices.pdf | 根据卷积核相似性进行剪枝]] 类似,都是考察卷积核的可替代性,可以用其他的卷积核包含的信息来替代某个卷积核,那么该卷积核即可被删去且对结果影响寥寥。
右上角即为传统方法进行剪枝,右下角为作者提出的方法(FPGM)进行剪枝
和之前看的 [[Network Compression Based on Filter Similarity Pruning for Fast Inference on Edge Devices.pdf | 根据卷积核相似性进行剪枝]] 类似,都是考察卷积核的可替代性,可以用其他的卷积核包含的信息来替代某个卷积核,那么该卷积核即可被删去且对结果影响寥寥。
欧式距离即两点之间的距离== 在两篇论文中,欧式距离都被用来进行计算并作为相关依据,该论文计算在欧式空间中的中位值来选择可被替代剪枝的卷积核,在根据卷积核相似性剪枝的论文中,通过计算每个卷积核之间的欧式距离,来作为衡量两者相似性的依据
主要贡献:
- 分析了之前的基于范数的剪枝规则,说明了其限制条件
- 提出了基于几何中位数进行剪枝的方法 FPGM
- 通过实验证明了 FPGM 的有效性
公式
首先,假设给定 n 个点 $a^{(1)},…,a^{(n)}$,$\forall a^{(i)} \in \mathbb{R}^d$,找到一个点 $x^$ 使得与所有其他点的欧氏距离之和最小$$x^ \in \mathop{argmin}{x \in \mathbb{R}^d}f(x)\quad where\quad f(x) \mathop=^{def} \sum{i \in [i,n]} ||x-a^{(i)}||_2 \tag{1}$$
argmin 指使后面这个式子达到最小值时的 x 的取值 $||x||_2$ 表示 x 的范数,实际就是模长的开根号,|x|表示模长,||x||表示在外套个绝对值 设$x=<x_1,x_2,x_3>$,则$||x||_2 = \sqrt{x_1^2 + x_2^2 + x_3^2}$ $\mathbb{R}^d$表示都是x都是d维的
接下来定义 $F_i^{GM}$ 为卷积核的第 $i$ 层的几何中位数,GM 指 Geometry Median - 几何中位数,$N_i$ 表示第 $i$ 层,也可表示输入维度,$N_{i+1}$ 表示第 $i+1$ 层,也表示输出维度 $$F_i^{GM}\in\mathop{argmin}{x \in\mathbb{R}^{N_i \times K \times K}}g(x) \tag{2}$$ 其中 $$g(x)\mathop{=}^{def}\sum{j’\in[1,N_{i+1}]}||x-F_{i,j’}||2 \tag{3}$$ 这里将 $x$ 点换成了卷积核,卷积核有 $N_i$ 个,每个大小为 $K \times K$ ,2、3 式即表示卷积核的几何中位数( $F_i^{GM}$ )为使得到该 $i$ 层每个卷积核的欧氏距离之和最小的一个值。 然后在第 $i$ 层中,找到离该几何中位数最接近的一个卷积核,将其删去,设为 $F{i,j^}$ 则 $$F_{i,j^}\in\mathop{argmin}{j’\in[1,N{i+1}]}||F_{i,j’}-F_i^{GM}||_2 \tag{4}$$
在接下来,作者做了一个简化,即”假设所有剪去的卷积核都在几何中心“,也就是 $$||F_{i,j^}-F_i^{GM}||2 = 0 \tag{5}$$ 这样的话也就是 $F{i,j^}==F_i^{GM}$,那么找到每个距离几何中位数最近的卷积核,就可以转成计算每个位于几何中心的卷积核,也就是这些卷积核全部满足(2)式的要求,即$$F_{i,j^}\in\mathop{argmin}_{j^\in[1,N_{i+1}]}g(x) \tag{6}$$
伪代码

实现代码
def get_filter_similar(self, weight_torch, compress_rate, distance_rate, length):
codebook = np.ones(length)
if len(weight_torch.size()) == 4:
filter_pruned_num = int(weight_torch.size()[0] * (1 - compress_rate))
similar_pruned_num = int(weight_torch.size()[0] * distance_rate)
weight_vec = weight_torch.view(weight_torch.size()[0], -1)
# norm1 = torch.norm(weight_vec, 1, 1)
# norm1_np = norm1.cpu().numpy()
norm2 = torch.norm(weight_vec, 2, 1)
norm2_np = norm2.cpu().numpy()
filter_small_index = []
filter_large_index = []
filter_large_index = norm2_np.argsort()[filter_pruned_num:]
filter_small_index = norm2_np.argsort()[:filter_pruned_num]
# distance using numpy function
indices = torch.LongTensor(filter_large_index).cuda()
weight_vec_after_norm = torch.index_select(weight_vec, 0, indices).cpu().numpy()
# for euclidean distance
similar_matrix = distance.cdist(weight_vec_after_norm, weight_vec_after_norm, 'euclidean')
# for cos similarity
# similar_matrix = 1 - distance.cdist(weight_vec_after_norm, weight_vec_after_norm, 'cosine')
similar_sum = np.sum(np.abs(similar_matrix), axis=0)
# for distance similar: get the filter index with largest similarity == small distance
similar_large_index = similar_sum.argsort()[similar_pruned_num:]
similar_small_index = similar_sum.argsort()[: similar_pruned_num]
similar_index_for_filter = [filter_large_index[i] for i in similar_small_index]
kernel_length = weight_torch.size()[1] * weight_torch.size()[2] * weight_torch.size()[3]
for x in range(0, len(similar_index_for_filter)):
codebook[
similar_index_for_filter[x] * kernel_length: (similar_index_for_filter[x] + 1) * kernel_length] = 0
print("similar index done")
else:
pass
return codebook